Case 2
Der findes flere forskellige typer af biosensorer, som anvender enzymer, antistoffer eller biomolekyler til at detektere det ønskede stof. Med Biotech Academys Biosensor kit kan du lave såkaldte “helcellebiosensorer”. En helcellebiosensor er en bakterie, eller en anden organisme, som kan detektere et input og respondere med et output. Det kan bruges i mange sammenhænge f.eks. medicinske eller miljømæssige. Man kan bruge mange forskellige organismer som vært til en helcellebiosensor som f.eks. gær eller E. coli. En type biosensor kan benyttes til at detektere forurenende stoffer såsom pesticider i et økosystem. En anden form for biosensor kan detektere riboflavin, som er et essentielt vitamin, i kroppen. Hvis du er interesseret i at lære, mere om biosensorer i den virkelige verden kan du læse mere på hjemmesiden (her).
I Biotech Academys Biosensor Case 2 øvelse skal du lave en helcellebiosensor som kan detektere stoffet acetylsalicylsyre. Biosensoren vil blive blålilla, når du tilsætter acetylsalicylsyre til mediet, hvor Biosensorbakterien vokser. Acetylsalicylsyre er det aktive stof i smertelindrende medicin som Kodimagnyl og Treo. Øvelsen har til hensigt at lære dig om transformation af bakterier, samt give dig praktisk erfaring med at arbejde med GMO-organismer og de sikkerhedsforanstaltninger, der hører med. Du vil også få en forståelse for biologiens centrale dogme, altså forholdet mellem DNA, mRNA og protein.
Hvis du ønsker at læse teorien som en pdf kan du downloade teorien her (opdateret d. 30/11 2020).
Her kommer et kort resumé af den teori, som du skal bruge for at forstå Biosensor Case 1 øvelsen. Vi har markeret de vigtige begreber med fed, så vær opmærksom på, om du har styr opå dem. Som du måske ved, er der forskel på hvordan prokaryote (bakterier) og eukaryote (planter, svampe og dyr) organismer fungerer. I følgende opfriskning snakker vi kun om prokayoter, da du i Biosensorøvelsen skal arbejde med en bakterie.
Vi begynder med det centrale dogme, som starter med, at DNA oversættes til mRNA. Denne proces kaldes transskription og varetages af mange forskellige proteiner bl.a. RNA-polymerasen. Næste skridt er en proces der ud fra mRNA-strengen, laves et protein. Denne proces kaldes translation og varetages af bakteriens ribosomer, der ved hjælp af tRNA-molekyler sammensætter proteinet, aminosyre for aminosyre, med mRNA som skabelon, indtil det færdige protein frigives fra ribosomet til bakteriens cytoplasma.

Gener er de områder i bakteriens DNA som, gennem det centrale dogme, bliver oversat til protein. De fleste gener i prokayoter har den samme overordnede opbygning: en promoter, et ribosomalt bindingssted, en kodende sekvens og en terminator. Figur 1 viser opbygningen af et typisk gen i prokayoter. Her kommer en lille oversigt over, hvad de enkelte genelementers funktion er:
- Promoteren er det sted hvor RNA-polymerasen binder til DNA-strengen og påbegynder syntesen af mRNA-strengen. Der findes forskellige typer af promotere. Nogle er altid aktive, dvs. at proteinet, som dette gen koder for, hele tiden vil blive produceret. Andre promotere bliver reguleret, og er dermed kun aktive under bestemte forhold, eksempelvis ved den rette temperatur eller under tilstedeværelsen af et bestemt molekyle. Regulering af promotere lærer du mere om, hvis du laver Biosensor Case 2 øvelsen.
- Ribosimalt bindingssted (RBS) er det sted, hvor ribosomet binder til mRNA- strengen og begynder at syntetisere proteinet, som genet koder for. Translationen stopper, når ribosomet når frem til et stopcodon, som er det sidste codon i den kodende sekvens (se næste punkt).
- Den kodende sekvens er den sekvens af DNA nukleotider, som dikterer sekvensen af mRNA-strengen og dermed proteinets aminosyresammensætning og rækkefølge. Det er altså den kodende sekvens, som bestemmer, hvilket protein genet koder for. Den kodende sekvens starter med et startcodon, ATG, hvor der indsættes aminosyren methionin ( forkortet Met eller M) og slutter med et stopcodon, TAG, TAA eller TGA, hvor der ikke indsættes nogen aminosyre. Det færdige protein kommer således til at bestå af den aminosyresekvens, som DNA-sekvensen fra startcodon til stopcodon koder for.
- Terminatoren stopper transskriptionen ved at vise RNA-polymerasen, at genet er slut. Når RNA-polymerasen når frem til terminatoren slipper den DNA-strengen og frigiver mRNA-strengen til bakteriens cytoplasma.
Det kan godt være svært at forstå, hvilke genelementer som hører til transskriptionen, og hvilke som hører til translationen. Man kan huske det ved, at de yderste genelementer, promoteren og terminatoren, bruges i det første skridt i det centrale dogme, nemlig transskriptionen. De inderste genelementer, det ribosomale bindingssted og den kodende sekvens bruges under translationen, som er det andet skridt i det centrale dogme.
DNA findes i forskellige former eksempelvis som lange kromosomer, der indeholder størstedelen af organismens gener. Bakteriers kromosomer er ofte et langt ringformet stykke DNA som er tilfældigt foldet over tusind gange i cellens cytoplasma. I modsætning til dette har eukaryoter kromosomer, som ofte er lineære DNA-stykker som er præcist viklet op omkring histonproteiner. Udover kromosomet indeholder bakterier plasmider, som er kortere ringformede stykker DNA, hvor bakterierne også har gener. Når man vil indføre et gen til en bakterie, er det ofte en fordel at indsætte genet i et plasmid og derefter indsætte plasmidet i bakterien.
Ligesom bakteriens kromosom skal plasmider replikeres for at de kan blive givet videre til bakteriens datterceller, når bakterien deler sig. Dette forgår ved en proces som hedder DNA-replikation. Replikationsprocessen starter ved origin of replication (ori) som er et genelement, som findes på plasmidet. ori giver bakterien mulighed for at replikere plasmidet, fordi det fungerer som startsted for DNA-polymerasen. Her kan DNA-strengen åbne op og lade bakteriens replikationsmaskineri (DNA-polymerase, helicase, primase osv.) begynde replikationen af plasmidet. Det betyder, at når plasmidet er inde i en bakteriecelle, vil bakterien kopiere plasmidet. Hvis du kunne tænke dig at få genopfrisket, hvordan DNA-replikationen fungerer, kan du se Biotech Academys Biostriben video her her.
Forhåbentligt virkede det ovenstående resume bekendt. Vi har lavet en liste over emner og begreber som er gode at have styr på inden du laver Biosensorøvelsen:
- Det centrale dogme.
- Kendskab til E. coli bakterier som modelorganisme.
- Plasmider
- Varmeshock transformation
- Genelementer: Promoter, ribosomalt bindingsted (RBS), den kodende sekvens, terminator og replikations origin (ori)
- Genregulering
- Antibiotika og brugen af antibiotika som selektionsmarkør
Disse emner bør du kunne læse om i din Bioteknologibog eller kigge på Biotech Academys projekt “Moderne genteknologi”, som du kan finde her.
Biotech Academys Biostriben har også tidligere lavet videoer som forklarer nogle af emnerne. Dem kan du finde her:
I Biosensor Case 2 skal du indsætte nogle gener i en E. coli bakterie. Der findes forskellige måder at indsætte DNA i bakterier.
Først og fremmest kan man indsætte en DNA-sekvens direkte i bakteriens kromosom. Alternativt, kan man benytte sig af et plasmid der bærer DNA-sekvensen.
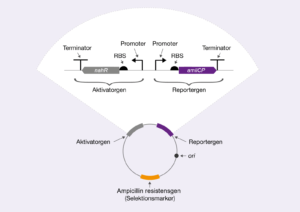
I denne øvelse skal du bruge et plasmid som vi kalder Case 2 plasmidet, hvor på der ligger tre gener: et reportergen, et aktivatorgen og et ampicillinresistensgen. Du kan se et plasmidkort over Case 2 plasmidet på figur 2.
Reportergenet
Vi starter med at tage et kig på reportergenet. Ligesom andre gener i bakterier består reportergenet af flere genelementer: en promoter, et ribosomalt bindingssted, den kodende sekvens og en terminator (se figur 2). Den kodende sekvens koder for et blålilla farveprotein AmilCP. Når det produceres bliver bakterierne blålilla. Promoteren i reportergenet er special, da RNA-polymeraser ikke kan starte transskription fra denne promoter uden at få hjælp fra aktivatorproteinet NahR sammen med acetylsalicylsyremolekyler.
Derfor vil det blålilla farveprotein som udgangspunkt ikke blive produceret, når der ikke er acetylsalicylsyre i mediet. Vi ser nærmere på dette senere.
Aktivatorgenet
Som du måske ved, er det ikke alle promotere, som altid er aktive. Bakterier og andre organismer bruger ændringer i promoteraktiviteten til at styre, hvornår de skal producere forskellige proteiner.
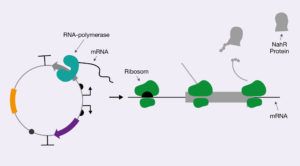
En E. coli bakterie har enzymer, som kan nedbryde og udvinde energien fra mange forskellige sukkerarter, f.eks. glukose og laktose. Hvis der kun er glukose til stede i bakteriens omgivelse ville det være spild af energi at producere enzymer, der kan nedbryde laktose.
Derfor har E. coli bakterier et system som sørger for at enzymerne som nedbryder laktose kun bliver produceret, når der er laktose i deres omgivelser. Dette system har du måske lært om i din bioteknologiundervisning, hvis du har lært om, det såkaldte lac-operon. Bakterier har mange lignende systemer, som der sørger for, at det kun er essentielle proteiner, der bliver udtrykt på et givet tidpunkt. Overordnet kaldes dette genregulering og er utrolig vigtigt for alle organismer så de kan bruge deres energi effektivt. Der findes mange forskellige mekanismer, der kan aktivere eller deaktivere gener på baggrund af alle mulige forhold, f.eks. tilstedeværelse af bestemte sukkerarter, temperatur og saltindhold. Det er bl.a. disse naturlige mekanismer, der kan udnyttes når man laver en biosensor.
I Biosensor Case 2 øvelsen skal du som sagt lave en biosensor, som reagerer på stoffet acetylsalicylsyre. I din biosensor vil genreguleringen ske ved et samspil mellem aktivatorgenet og reportergenet. Aktivatorgenet består ligesom reportergenet af fire genelementer: en promoter, et ribosomalt bindingssted, den kodende sekvens og en terminator (se figur 2). Den kodende sekvens koder for proteinet NahR, som er en aktivator for promoteren, som findes i reportergenet. NahR bliver udtrykt af en konstitutiv promoter, det vil sige at den altid er “tændt”. Altså producerer biosensorbakterien hele tiden NahR-proteinet (se figur 3). Umiddelbart skulle man tro, at reportergenet dermed også hele tiden ville blive udtrykt, men sådan forholder det sig ikke. Det skyldes at NahR skal binde acetylsalicylsyre for at kunne aktivere reportergenet. Man siger at, acetylsalicylsyre fungerer som en inducer til NahR. Da bindingen af acetylsalicylsyre “tænder” aktivatorproteinet NahR ved at ændre dets form, så det kan binde RNA polymerasen til promoteren.
Reguleringsmekanismen
Reguleringsmekanismen i acetylsalicylsyre biosensoren er ikke fuldt ud opklaret, men der eksisterer en velbegrundet teori om, hvordan den virker. Mellem aktivator- og reportergenet findes fire kopier af en DNA-sekvens, som NahR-proteinet kan binde til (se figur 4.1).
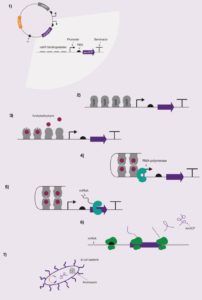
Udover at NahR-proteinet kan binde til DNA, kan det også binde til acetylsalicylsyre. Man mener at binding af acetylsalicylsyre medfører en konformationsændring af NahR-proteinet, som gør, at det kan binde til andre NahR-proteiner og dermed indgå i en kvartenær proteinstruktur.
Mange forskere mener at et NahR-protein binder til hver af de fire NahR-bindingssekvenser, som findes mellem aktivatorgenet og reportergenet (se figur 4.2).
Dernæst binder de fire NahR-proteiner hver til et acetylsalicylsyremolekyle. Dette gør at NahR-proteinerne skifter til en anden konformation (se figur 4.3).
Nu folder DNA-strengen rundt og gør det muligt for de fire NahR-proteiner at interagere to og to (se figur 4.4).
Når NahR-proteinerne er bundet sammen, kan de hjælpe RNA-polymerasen med at binde til promoteren og påbegynde transskriptionen (se figur 4.5). mRNA-strengen kan nu translateres til AmilCP, og bakterien vil blive blålilla (se figur 4.6 og 4.7).
For at opsummere: RNA-polymerasen skal bruge NahR, som sidder sammen to og to, for at kunne begynde transskriptionen. NahR skal bruge acetylsalicylsyre for at kunne sætte sig sammen to og to. På den måde bliver reportergenet kun udtrykt, når der er acetylsalicylsyre i bakteriens omgivelser.
Biologiske processer er meget sjældent stringente. Det samme gælder for reguleringsmekanismen i denne biosensor. Det betyder, at selv om RNA-polymerase i teorien ikke burde kunne transskribere reportergenet, når der ikke er acetylsalicylsyre til stede, sker det stadigvæk. Der bliver dog ikke produceret lige så meget af det blålilla farveprotein, som når der er acetylsalicylsyre til stede. Dette fænomen kaldes, at promoteren er utæt.
Derfor kan du, når du laver biosensoren i laboratoriet, godt opleve, at bakteriekolonierne vil være svagt farvede, allerede inden du har udpladet bakterierne på, medium med acetylsalicylsyre.
Selektionsmarkør
Vi har nu beskrevet funktionen af både aktivator- og reportergenet. Det sidste gen, som findes på Case 2 plasmidet, koder for ampicillinresistens. Fra dette gen bliver der produceret et protein, som gør bakterien i stand til at overleve selv om der er ampicillin antibiotikummet i dens omgivelse. Genet for resistens kaldes en selektionsmarkør, da det muligører at selektere (udvælge) de bakterier, som har optaget biosensorplasmidet frem for de bakterier, som ikke har. En selectionsmarkør er særlig brugbar, hvis dit indsatte gen ikke giver bakterien en anden fænotype (for eksempel farve). Resistensmarkøren gør det nemmere, at arbejde videre med den genmodificerede bakterie. I Case 2 vil din biosensor ikke have nogen farve, før den bliver aktiveret af acetylsalicylsyre. Selektionsmarkøren gør det derfor nemt for dig at udvælge de bakterier, der har optaget Case 2 plasmidet.
Origin of replication
På Biosensor Case 2 plasmidet findes også et replikations start sted (ori), der bliver genkendt af bakteriens DNA-polymeraser, og som starter DNA-replikationen af plasmidet her. Således bliver der lavet flere kopier af biosensorplasmidet inde i bakterien. Når der er flere kopier af plasmidet, vil der altså være flere kopier af genet for det rødt fluorescerende protein. Det betyder, at flere RNA-polymeraser samtidigt kan transskribere genet, så der bliver produceret mere af det blålilla farveprotein AmilCP. Forskellige ori kan give forskellige kopital (det gennemsnitlige antal kopier af et plasmid i hver bakterie). Nogle ori giver kun få kopier (1-10 plasmider pr. bakterie), mens andre, som f.eks. det i Biosensor Case 2 plasmidet, er blevet estimeret til at give 200-300 kopier i hver bakterie.
Videoen nedenfor giver en oversigt over plasmidets opbygning.
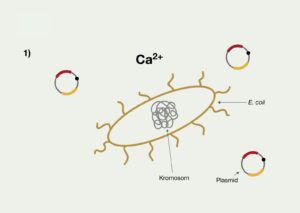
Optagelsen af fremmed DNA i en bakterie kaldes transformation.
Metoderne for, hvordan transformationen udføres, varierer mellem forskellige bakterier. I Biosensor Case 2 øvelsen skal du indsætte et plasmid (Case 2 plasmidet) i en E. coli bakterie.
OBS: Den følgende beskrivelse af transformationen tager udgangspunkt i Case 1 plasmidet, dette betyder at transformanterne vil være røde når E.coli har optaget plasmidet, men teorien bag transformationen er den samme.
For at udføre transformationen skal du anvende varmeshock transformationsmetoden, hvor du bruger kalciumchlorid. Med denne metode behandles bakterierne først med kalcium-ioner (Ca2+), for at gøre dem kemisk kompetente (se figur 5.1).
Dernæst tilsættes plasmidet til de kemisk kompetente bakterier og de udsættes for et varmeshock, som resulterer i, at plasmidet bliver optaget i de mest kompetente bakterier (se figur 5.2). De bakterier som har optaget biosensorplasmidet kaldes transformanter.
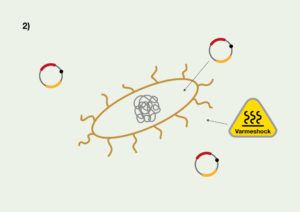
Nu har du en blanding af bakterier som har optaget Case 2 plasmidet (transformanter) og bakterier som ikke har optaget plasmidet. Sidstnævnte kaldes vildtypebakterier, fordi de ikke har optaget nogle nye gener og altså har den oprindelige genotype. Denne blanding kalder vi for transformationsblandingen.
Mekanismen for hvordan varmeshocket virker, er endnu ikke opklaret. Man har blot fundet ud af at øge transformationseffektiviteten, altså hvor mange af de kompetente bakterier som optager plasmidet.
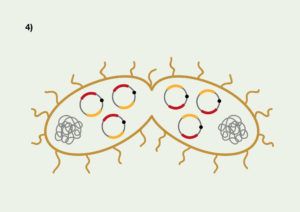
Transformanterne begynder nu at replikere biosensorplasmidet via ori (se figur 5.3). Efter varmeshocket tilsættes LB-medium, som er et nærings- og energiholdigt vækstmedium. Næringen bruger alle bakterierne (både transformanterne og vildtypebakterierne) til at vokse og dele sig (se figur 5.4).
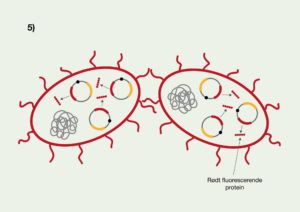
Transformanterne begynder også at syntetisere det rødt fluorescerende protein og ampicillinresistensproteinet, ud fra generne på Case 2 plasmidet (se figur 5.5).
Det tager dog lang tid før der er produceret nok rødt fluorescerende protein til, at det kan ses med det blotte øje.

Næste skridt er at selektere (udvælge) transformanterne.
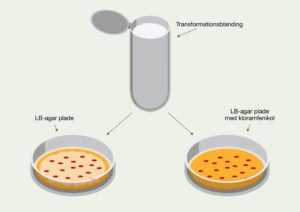
Det gøres ved at udplade transformationsblandingen på en LB-agarplade med ampicillin tilsat. Transformanterne kan overleve, fordi de udtrykker ampicillinresistensproteinet, mens vildtypebakterierne vil blive dræbt af ampicillinen.
Hvis transformationsblandingen blev udpladet på en LB-agar plade uden ampicillin, ville der vokse en blanding af transformanter og vildtypebakterier på LB-agarpladen. Det ville gøre det svært at udvælge transformanterne til videre arbejde (se figur 6). Selektionmarkører er derfor særligt smarte, hvis genet ikke gør det nemt for dig at se forskel på bakterier, hvor transformationen er lykkedes og hvor den ikke er.
Transformation af bakterier i laboratoriet from Biotech Academy on Vimeo.
Efterhånden som mulighederne udvides for at flytte rundt på gener og ændre dem, bliver det vigtigt at tage etisk stilling til, hvor grænserne sættes. Genteknologien gør det muligt at arbejde kreativt med vores biologiske arv og potentielt skabe nye transgene organismer, der kan løse problemer med sundhed, sult eller manglende ressourcer. Men i nogle situationer kan genteknologi også medføre en vis risiko for uoprettelig spredning af fremmede gener i naturen. Man må derfor vurdere, om brugen er acceptabel, for eksempel hvis nytteværdien kan betyde, at der reddes liv, eller at prisen på dyrkningen af afgrøderne falder [1].
Ifølge lovgivningen skal genmodificerede planter godkendes i EU, før de kan udsættes og dyrkes i naturen. En ny gen-modificeret plante skal derfor risikovurderes af myndighederne, hvor der blandt andet bliver udført ekstra sikrede forsøgsdyrkninger. Planten kan så godkendes, hvis myndighederne vurderer, at den ikke udgør nogen risiko for miljøet, samt for dyrs og menneskers sundhed, dette kaldes også for forsigtighedsprincippet.
Et fremført argument imod gensplejsning af dyr og planter er, at det er et kunstigt, menneskeligt indgreb, som påvirker både deres integritet og naturlighed. Etisk taler man om, at naturen kan miste sit væsen og reduceres til en ren menneskelig ressource eller råstof, mens artsforskellene forstyrres, når transgene organismer skabes. Integriteten skades, når et dyr mister sin naturlige helhed og bringes ud af sin verden, som det måske har tilpasset sig gennem mange tusinde års udvikling og evolution [1]. Nogen opfatter brugen af gensplejsning som at ‘lege Gud’ og er derfor stærke modstandere. Hvis du ønsker at læse mere om bioetik, findes der blandt andet et etik interview med teolog Mickey Gjerris og professor Kasper Lippert-Rasmussen der har et meget forskelligt synspunkt på brugen af GMO [2].
I vurderingen af nye gensplejsede organismer anlægger flere en nytteetisk betragtning og mener derfor, at godkendelse af genmodificerede planter og dyr godt kan accepteres, hvis de tjener en tilstrækkelig nytteværdi.
[1] Jesper Wind. Moderne genteknologi. https://www.biotechacademy.dk/ undervisning/gymnasiale-projekter/moderne-genteknologi/, 2000.
[2] Det etiske råd. Etikinterview om gmo. http://www. etiskraad.dk/etiske-temaer/natur-klima-og-foedevarer/ undervisning-til-gymnasieskolen/gmo/etikinterview, 2011.
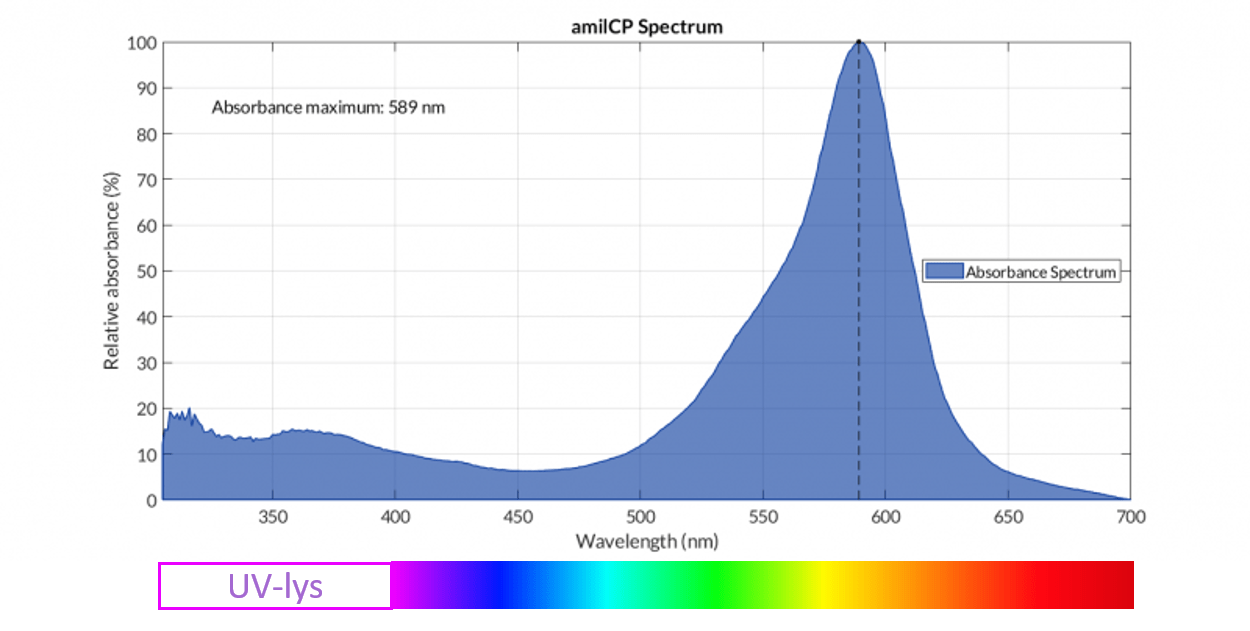
Reportergenet AmilCP, der findes i case 2 plasmidet, koder for et protein, der er isoleret fra koralen Acropora millepora. Deraf kommer proteinets navn, AmilCP, hvor CP står for chromoprotein. Ordet “chromo” kommer fra græsk og betyder farve.
AmilCP er således et farveprotein, der har en karakteristisk blålilla farve. Til forskel fra reportergenet i case 1, Rødt Fluorescerende Protein (RFP), kan farven fra AmilCP ses med det blotte øje, dvs. uden at skulle belyse bakterierne med UV-lys. Dette gør AmilCP og andre chromoproteiner velegnede til forskningsbrug.
Årsagen til den karakteristiske blålilla farve skyldes, at proteinet AmilCP absorberer en stor del af det synlige lys i bølgelængder, der svarer til rød, orange, gul og grøn, og reflekterer lys med bølgelængder, der svarer til blå og lilla (se figur 7 nedenfor).
Her finder I de øvelsesvejledninger der skal benyttes til Biosensor Case 2 øvelsen.
Biosensor Case 2 Øvelsesvejledningen skal benyttes af både underviser og elever:
Som underviser skal du også benytte følgende vejledninger:
Hvis du ønsker at lave både Case 1 og Case 2 på samme tid, kan du benytte nedenståede protokol:
Her finder I opgaver, der er lavet specifikt til Biosensor Case 2 øvelsen.
Her finder I flowsheet til Biosensor Case 2 øvelsen. Vi anbefaler at flowsheetet udfyldes før I går i laboratoriet.
Hvis du ønsker at lave både case 1 og case 2 med dine elever så har vi lavet et flowsheet til når du laver begge øvelser:
Her på siden kan du finde et dokument, der indeholder information omkring de tekniske deltaljer i Case 2 øvelsen. Dokumentet beskriver de forskellige elementer der indgår i Case 2 plasmidet såsom backbone samt indhold i reportergenet og aktivatorgenet, man kan selv vælge om man vil benytte dokumentet til undervisningen.
Indholdet i dette dokument er ikke noget eleverne bør vide for at forstå øvelsen. Det kan dog være interessant for den ekstra interessede elev at kigge nærmere på de forskellige elementer i plasmidet, og hvor de stammer fra.
Man kan downloade dokumentet her (opdateret d. 16/11 2020).
For at gøre det så let for eleverne at lave Biosensor Case 1 øvelsen har vi lavet en række videoer der viser: korrekt pipettering, udpladning af bakterier og renstrygning af bakterier. Disse videoer kan du finde her.
